geo数据库怎么弄?其实也没那么复杂,跟着步骤慢慢来就好啦
- 问答
- 2025-12-24 18:28:10
- 2
“geo数据库怎么弄?其实也没那么复杂,跟着步骤慢慢来就好啦。” 这句话听起来像是朋友间的鼓励,确实,虽然“数据库”这个词听起来有点技术性,但GEO数据库的“弄”主要指的是从上面找到你需要的数据并成功下载分析,而不是让你从零搭建一个数据库,整个过程就像是在一个巨大的公共图书馆里,按照索引找到一本特定的书,然后把书里的内容复印下来带回家研究,下面我就一步步带你走一遍这个流程。

你得知道GEO数据库是什么,根据“NCBI GEO(基因表达综合数据库)官方介绍”,它是一个国际性的公共数据库,主要存放高通量的基因表达数据,全世界很多科学家做完基因测序之类的实验后,都会把原始数据和结果上传到这里,供其他人免费下载和再分析,你“弄”GEO数据库,99%的情况是去里面“淘金”,利用别人公开的数据来验证你自己的科学假设或者发现新的线索。
第一步,明确你的目标,你去图书馆总不能瞎逛吧?得先知道你想找什么主题的书,用GEO也是一样,在打开网站之前,先想清楚:我想研究什么疾病?肝癌”?还是想研究某种药物处理后的细胞变化?用紫杉醇处理乳腺癌细胞”?或者你对某个特定的基因感兴趣,想看看它在哪些条件下会发生变化?把这个目标用几个关键词写下来,这是你后续所有操作的指南针。
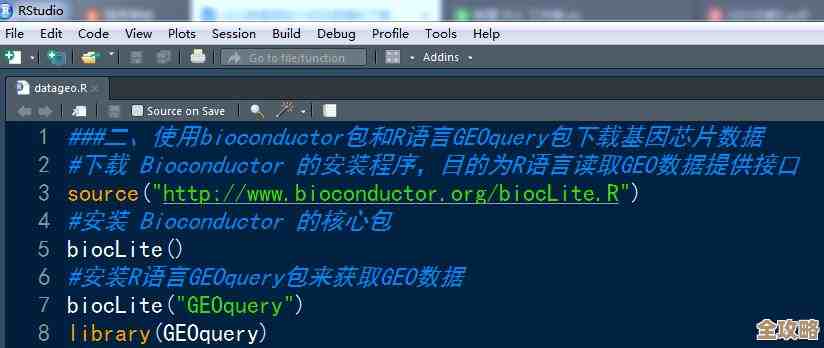
第二步,访问并搜索,GEO数据库的网址是很容易找到的,你可以在搜索引擎里直接搜“GEO数据库”,第一个结果通常就是美国国立生物技术信息中心(NCBI)旗下的GEO网站,点进去之后,你会看到一个搜索框,这时候,把你第一步想好的关键词输进去,liver cancer”(肝癌)和“Homo sapiens”(人类),直接搜可能会出来太多结果,这时候就要用到高级搜索技巧了,根据“很多生物信息学入门指南的建议”,你可以组合使用一些标签来精确搜索,你可以搜索 "liver cancer"[Title] AND "Homo sapiens"[Organism],这样就能把范围缩小到标题中包含肝癌并且研究对象是人的数据集,这一步可能需要一点耐心,多尝试几个关键词组合,直到找到看起来最符合你需求的数据系列(Series,简称GSE)。
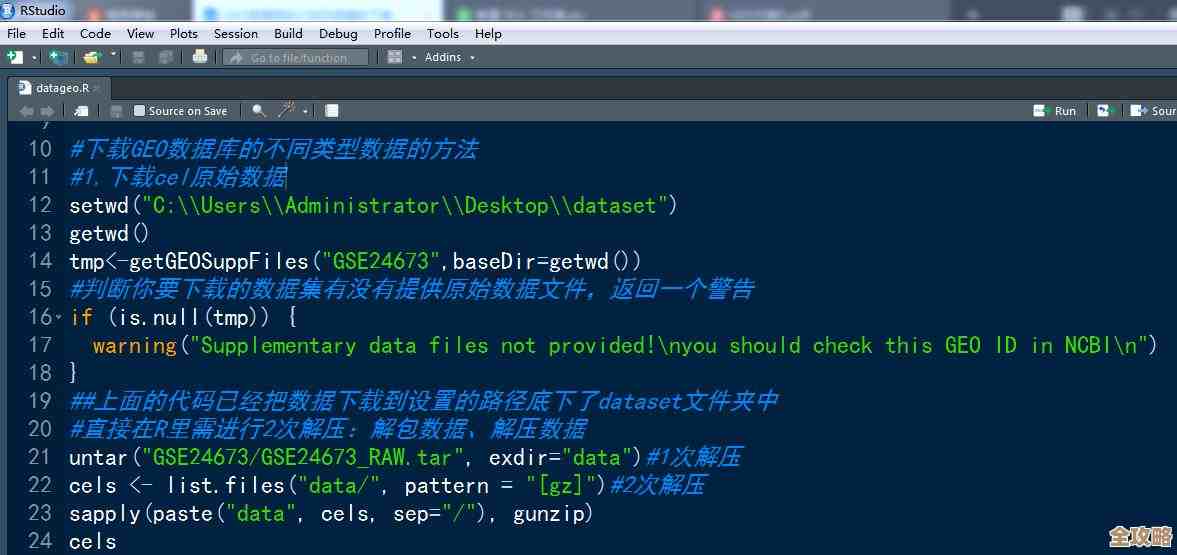
第三步,看懂搜索结果页,当你点击一个GSE编号(比如GSE12345)进入详情页时,可能会被密密麻麻的信息吓到,别慌,重点看这几块:1. 标题和摘要:快速判断这是不是你要的东西,2. 实验设计:看看他们用了多少样本,分了几组(比如正常组 vs 疾病组,对照组 vs 药物处理组),这是理解数据的基础,3. 数据类型:是mRNA测序数据还是芯片数据?这决定了你后续的分析方法,4. Supplementary file(补充文件)栏目:这是关键中的关键!你要下载的数据文件都在这里,对于芯片数据,通常会有一个包含所有样本表达量的整合表格文件(名字里常带有“series matrix”或“表达矩阵”),可能还有一个压缩包包含每个样本的原始数据,对于测序数据,可能会提供原始测序文件(如FASTQ格式)或已经处理好的计数矩阵(count matrix)。
第四步,下载数据,找到你想要的文件后,直接点击链接就可以下载了,表达矩阵文件(通常是.txt或.csv格式)一般不大,下载很快,但如果涉及到原始数据,文件可能会非常大(几个G甚至更大),需要确保你的网络稳定,并且电脑有足够的存储空间,浏览器直接下载大文件容易中断,可以参考“一些技术论坛上的经验”,使用一些支持断点续传的下载工具会更稳妥。
第五步,这才是真正“弄”数据的开始——数据分析,下载下来的原始数据你不能直接拿来就用,就像买回来的菜得先洗切炒一样,你需要用软件(比如R语言加上Bioconductor的一系列包,或者一些图形化工具)对数据进行处理,这个过程包括数据清洗(去掉质量不好的数据)、标准化(让不同样本之间的数据可以公平比较)等,之后,你就可以进行各种分析了,比如找差异表达的基因、做功能富集分析看看这些基因主要参与哪些生物过程、或者构建一些预测模型,这一步专业性稍强,但网络上有海量的免费教程和代码范例,完全可以跟着学。
保持耐心和探索精神,第一次接触GEO可能会觉得有点不知所措,这非常正常,不要指望一次就能完全掌握,你可以先从一个小而简单的数据集开始练手,比如找一个样本量少、实验设计清晰的数据集,成功走完从下载到简单分析的全程,建立信心,遇到问题时,善用搜索引擎,你碰到的问题很可能早就有人遇到过并且有解决方案了,GEO是一个宝库,只要你愿意花时间慢慢摸索,一定能从中获得对你研究有价值的信息,真的,没那么复杂,一步一步来就好啦。
本文由芮以莲于2025-12-24发表在笙亿网络策划,如有疑问,请联系我们。
本文链接:http://www.haoid.cn/wenda/67702.html